Le bêtisier du
séquenceur ...
Préface juridique: toute
ressemblance avec des séquences de personnes vivantes ou parties
est purement fortuite. D'ailleurs j'ai caché les noms pour
préserver votre anonymat.
Pour les signaux, les infos sont consultables à des endroits
variables selon le logiciel d'analyse. Cherchez ...
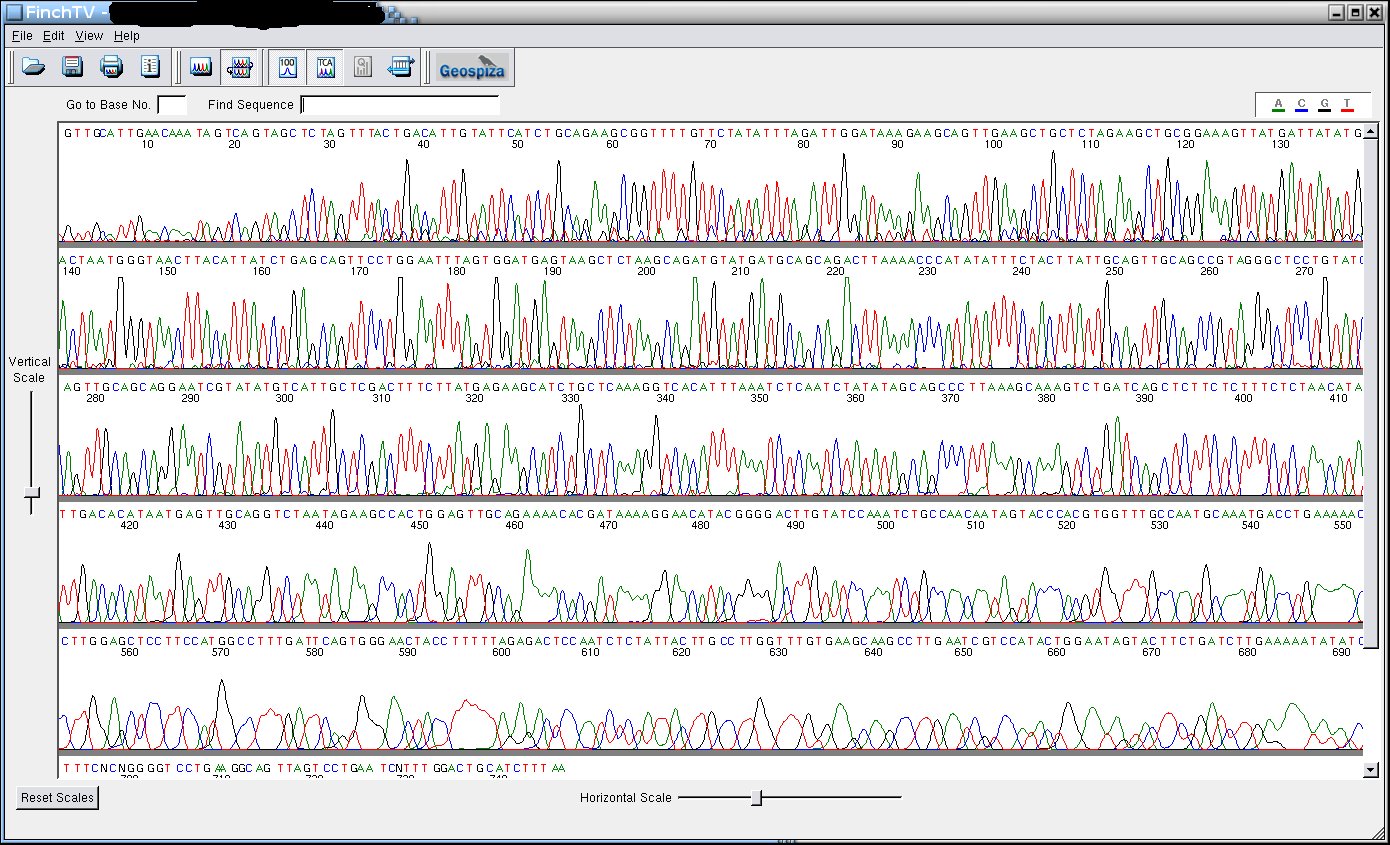
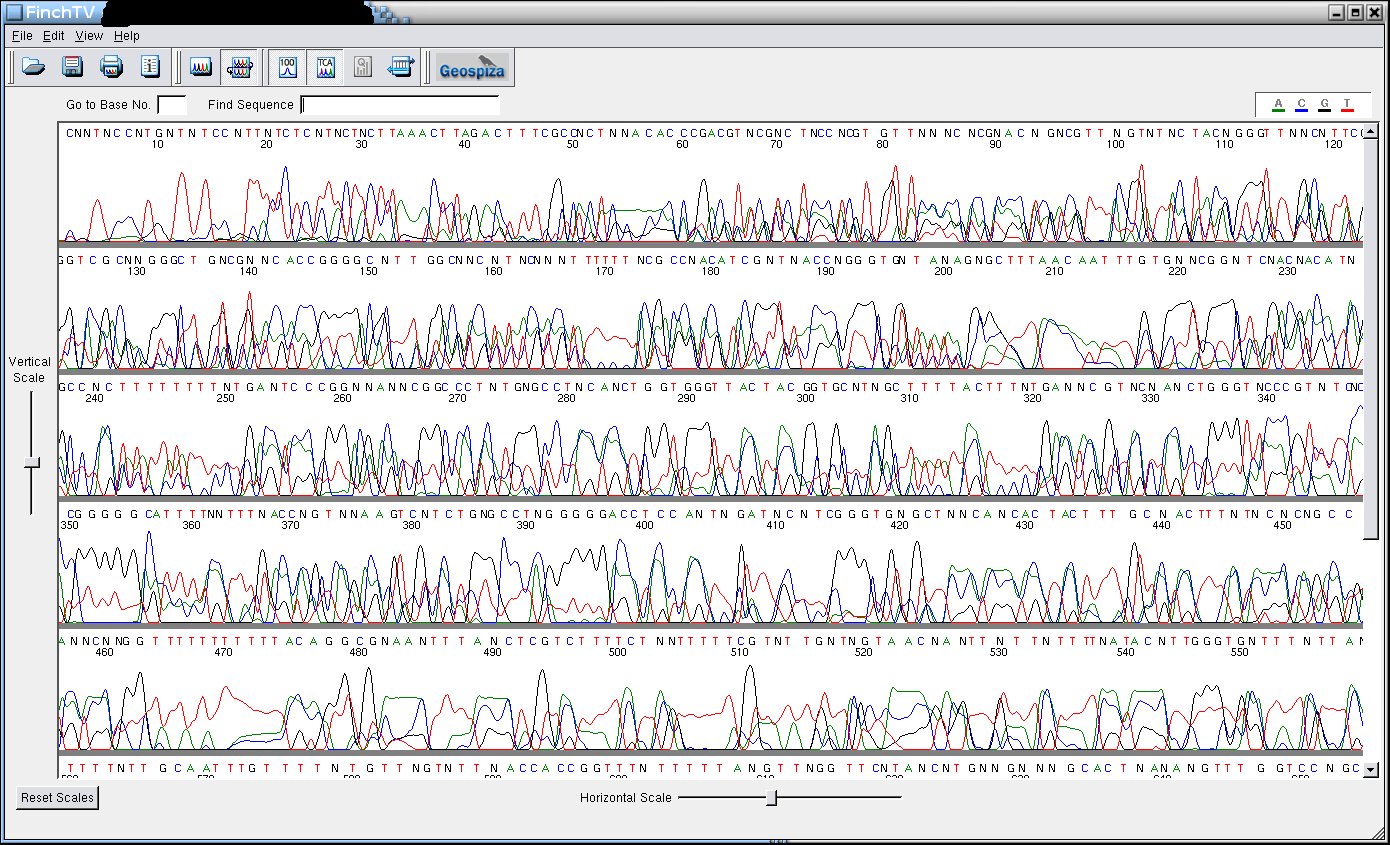
Quand ça marche
Ça ressemble à ça. Pas d'autres
commentaires. D'ailleurs, quand ça marche, personne ne vient me
voir ...
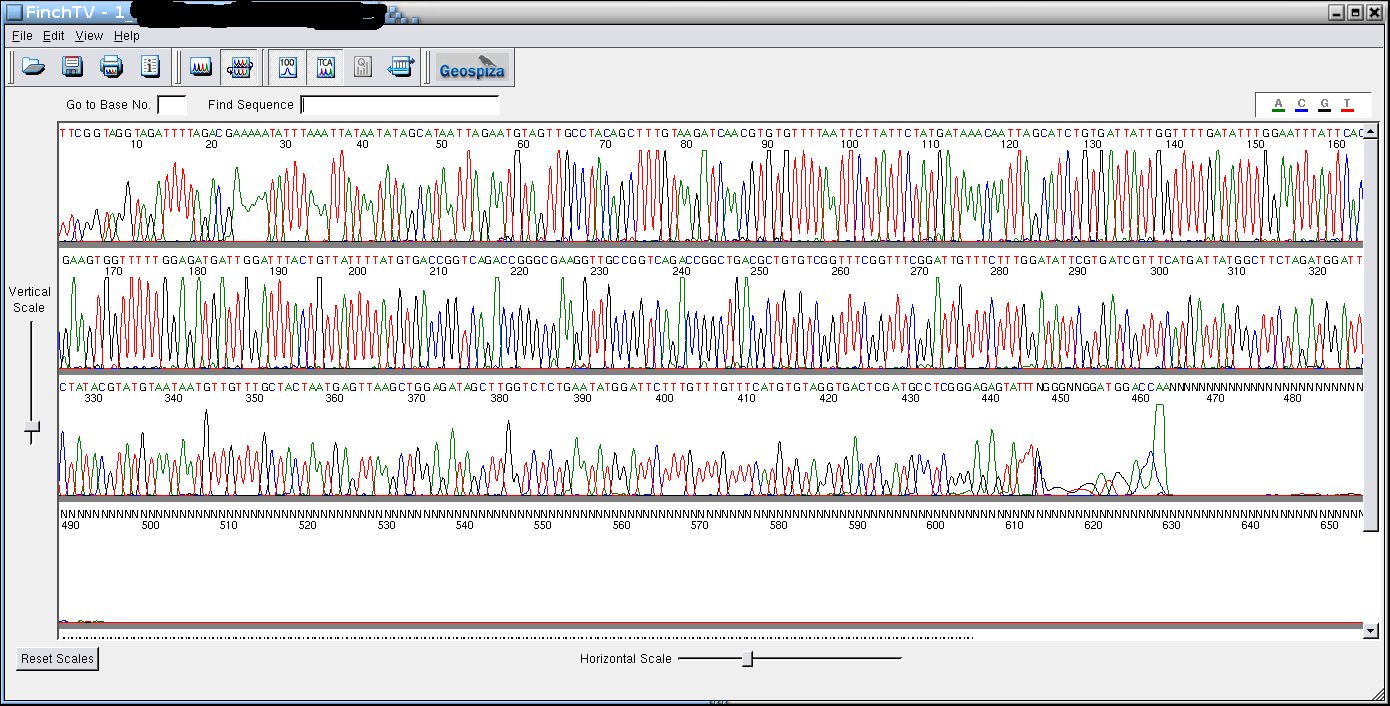
Les produits PCR
Ceux-ci se
séquencent bien d'habitude. Il est fortement conseillé de purifier et de doser les produits PCR. Souvent ça marche mieux avec
une amorce interne plutôt qu'avec une des amorces PCR. Il faut
bien doser les quantités et bien appliquer la règle de
trois dans le calcul de la quantité. Ne pas hésiter
à mettre très peu d'ADN pour de petits fragments. Les
séquences se terminent par un grand pic "A". S'il y a des restes
de la séquence analysée juste avant, on voit parfois de
petits pics correspondant à celle-ci après la fin du
fragment PCR.
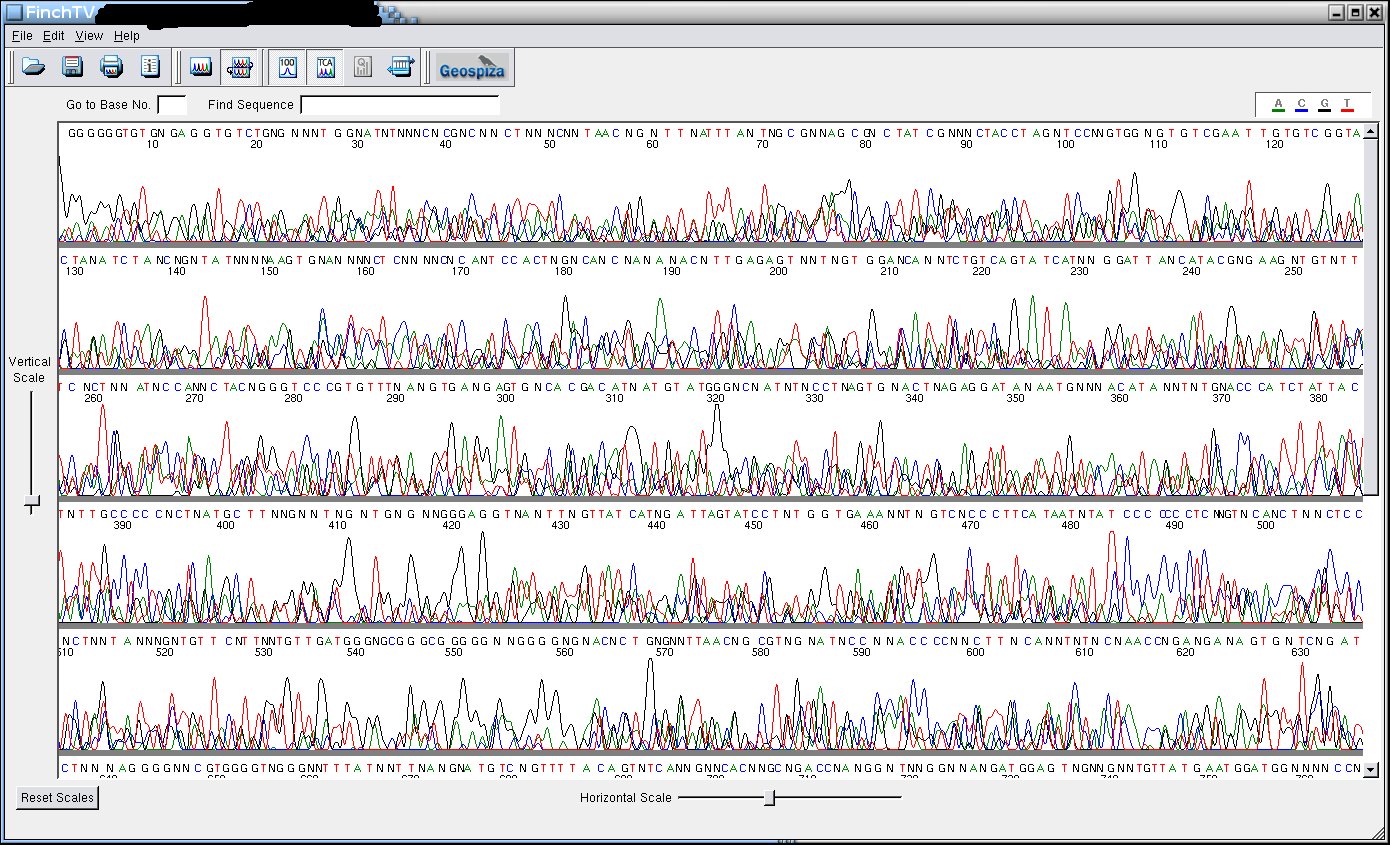
Quand
ça ne marche pas
Un peu n'importe quoi
Des pics un peu partout ou carrément l'encéphalogramme
plat. Il y a plusieurs possibilités. Le plus simple, c'est qu'il
n'y a pas, ou plutôt pas assez, d'ADN. De
préférence, dosez vos échantillons sur gel
d'agarose par rapport à un plasmide qui a bien marché en
séquençage. D'autres possibilités sont l'absence
de site pour votre amorce (voire absence d'amorce, ce qui arrive ...)
ou de l'ADN "sale". Il est conseillé de redissoudre son ADN dans
une solution faiblement tamponnée, du Tris-HCl 1mM, pH 8,5.
Évidemment, ne pas mettre d'EDTA: il y a du magnésium
dans le tampon de séquençage. D'autres produits inhibent
la réaction: phénol, éthanol, ARN, sels, ... Bref,
une préparation propre est la meilleure garantie d'une belle
séquence.
Méfiez-vous, si vous n'avez pas VOTRE séquence, vous
pouvez récupérer le fantôme de celle qui
était dans le capillaire juste avant!
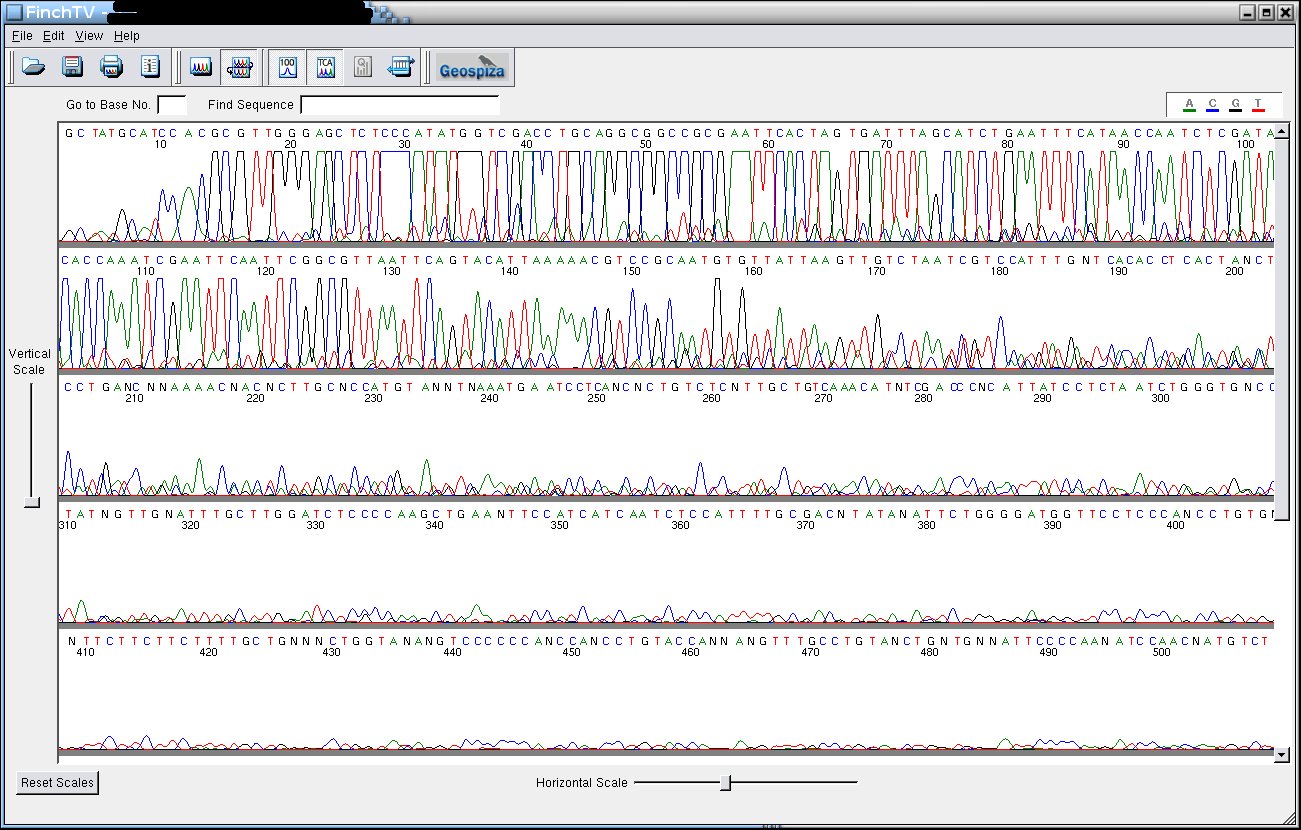
Trop d'ADN
La séquence
commence avec des pics au plafond, mais le signal baisse très
vite. Ça vient d'un mauvais rapport amorce/matrice, en
général parce qu'on a mis trop d'ADN, mais il peut y
avoir un problème de quantité d'amorce.
Beaucoup trop d'ADN
Ça peut
ressembler à "pas assez de matrice" mais les signaux sont
TRÈS forts (plusieurs milliers). Le phénomène se
manifeste d'abord par des "pics sous les pics" et dans les cas
extrêmes, par des pics écrasés. Redoser,
recommencer.
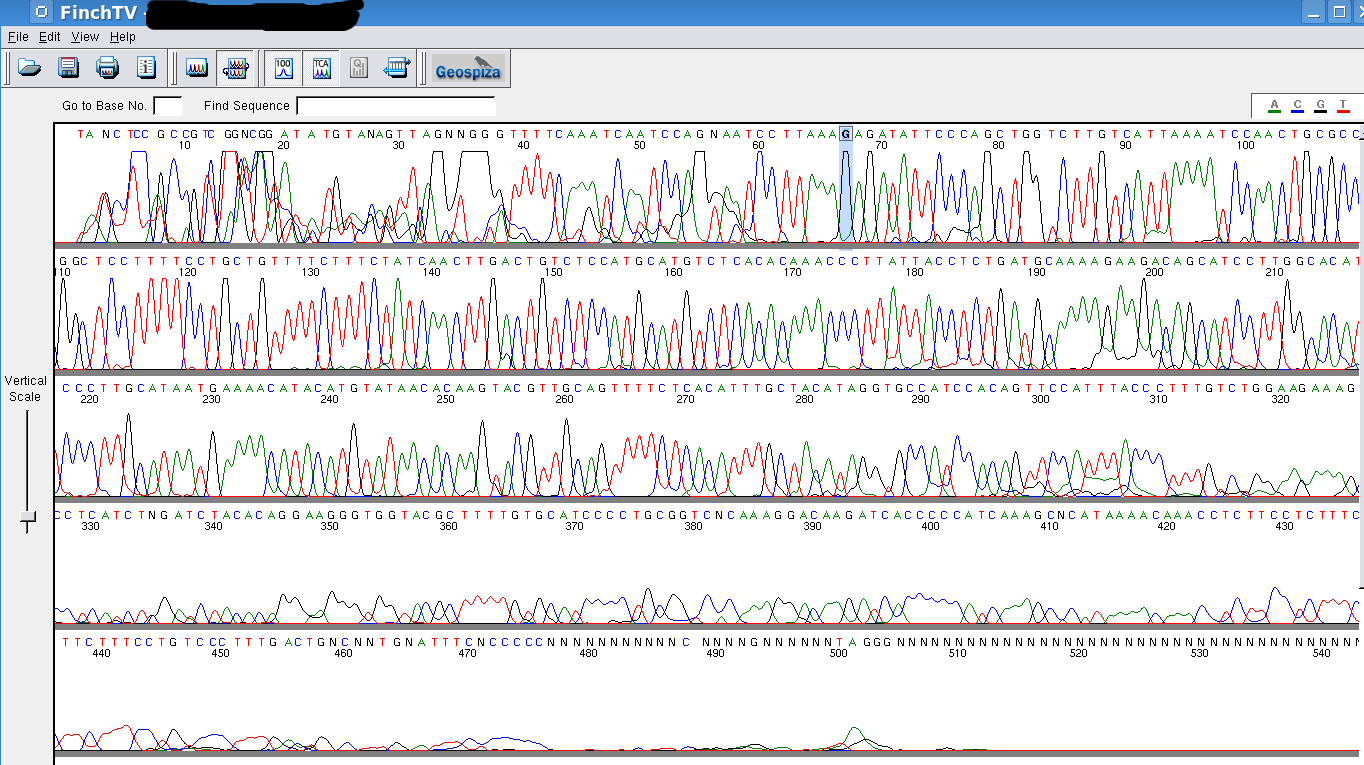
Le mélange de séquences
Il y a en fait
deux cas de figure. Si on voit les traces de deux séquences
dès le début, c'est en général un
problème d'amorce. Sur les produits PCR séquencés
directement, il ne faut pas qu'il reste des traces des deux amorces
utilisées pour l'amplification. Sinon, ça peut marcher (à peu près):
Comme ça peut donner ça:
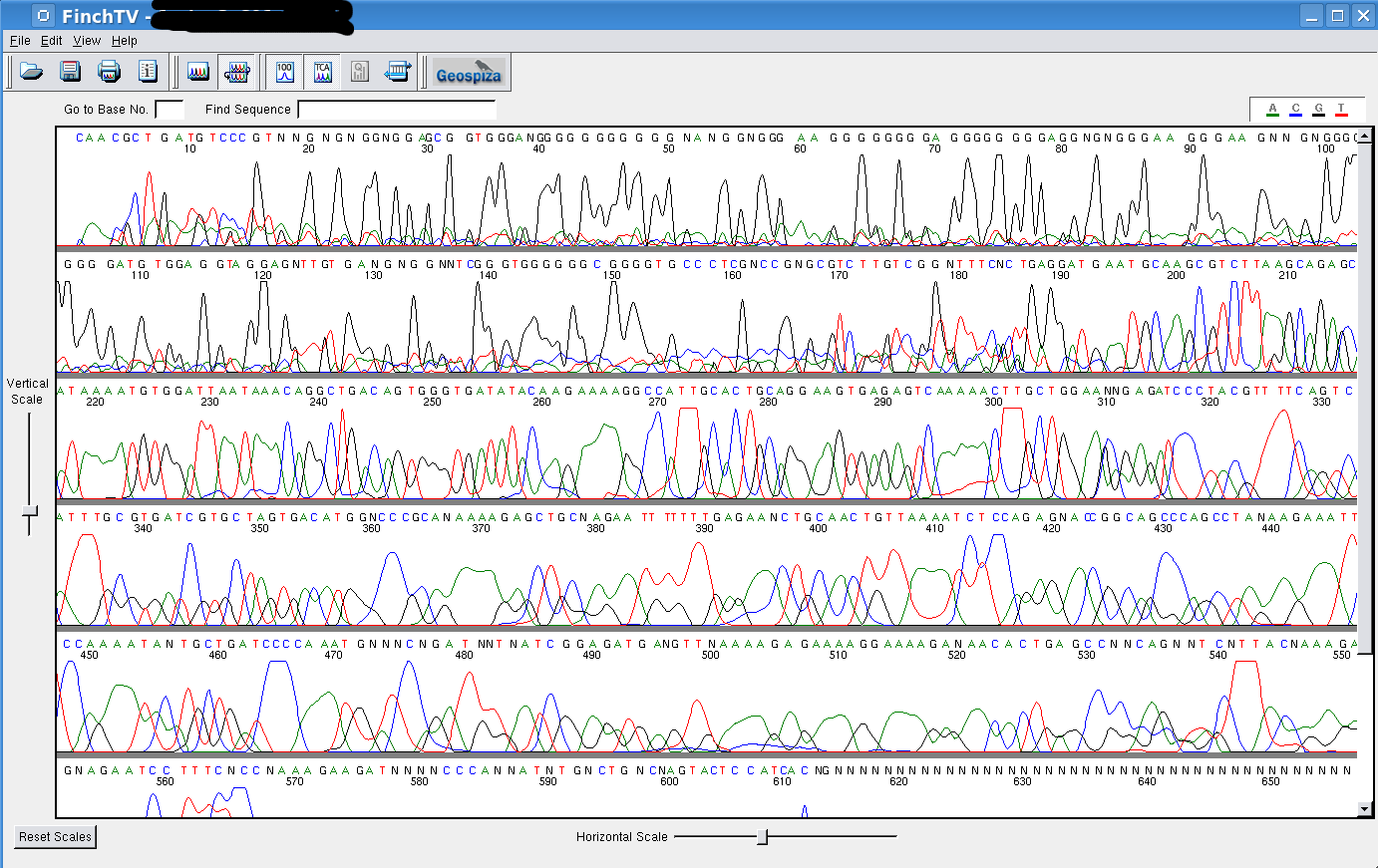
Autrement, ça vient de la
présence de deux sites d'amorçage sur la matrice. C'est
un problème surtout sur les grandes matrices.
Dans l'exemple donné ici, c'est un problème de
mélange de clones. La séquence se lit parfaitement tant
qu'on est dans le vecteur, mais on voit deux traces dès qu'on
passe le site de clonage. Ceci semble être un problème
avec certains clones pGEM-T, peut-être quand il y a trop de
produit PCR dans la ligation.
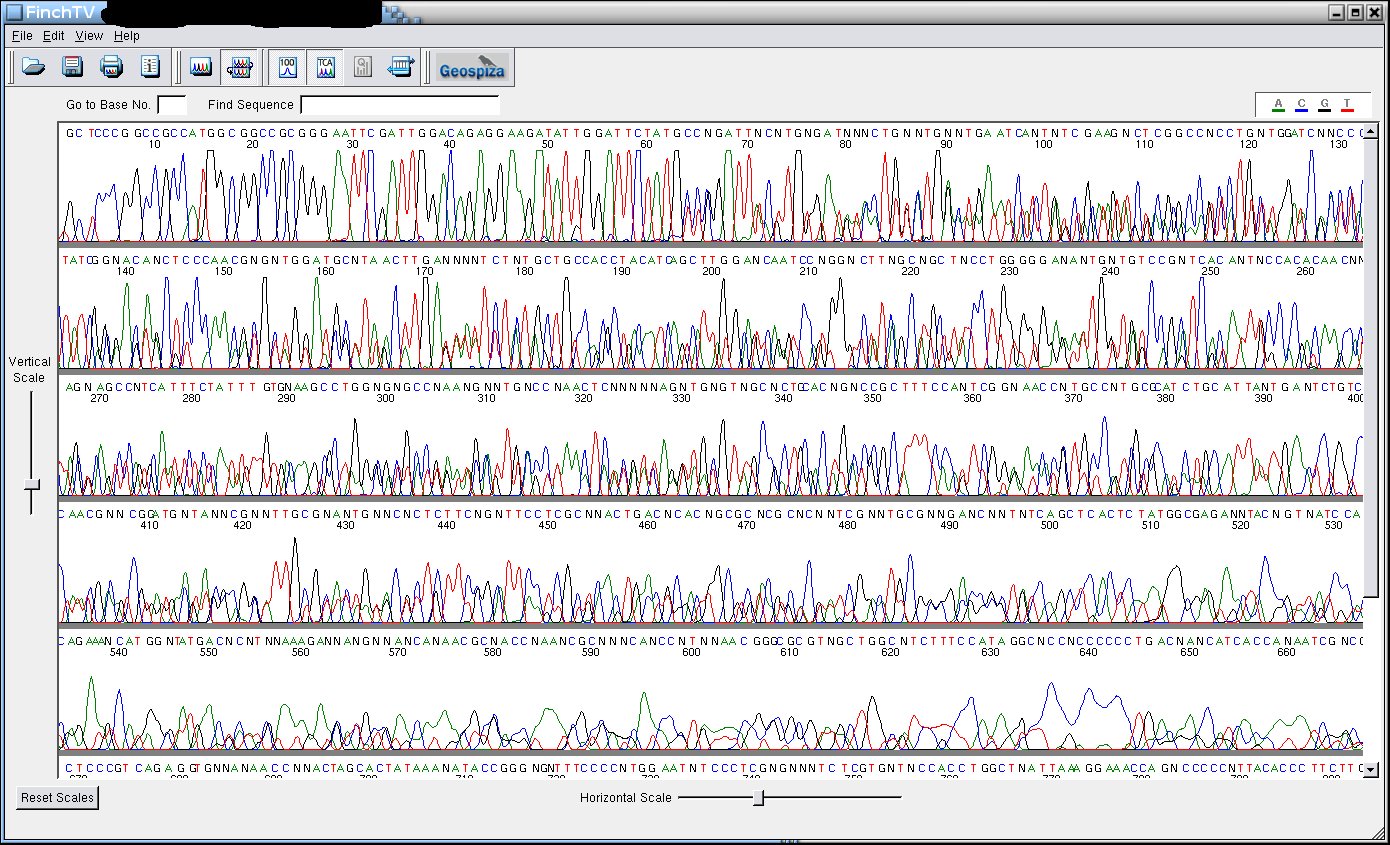
Contamination
La contamination
des preps par d'autres molécules, notamment par l'ADN
génomique de coli, provoque une migration en "vagues". Une seule
solution: recommencer la prep.
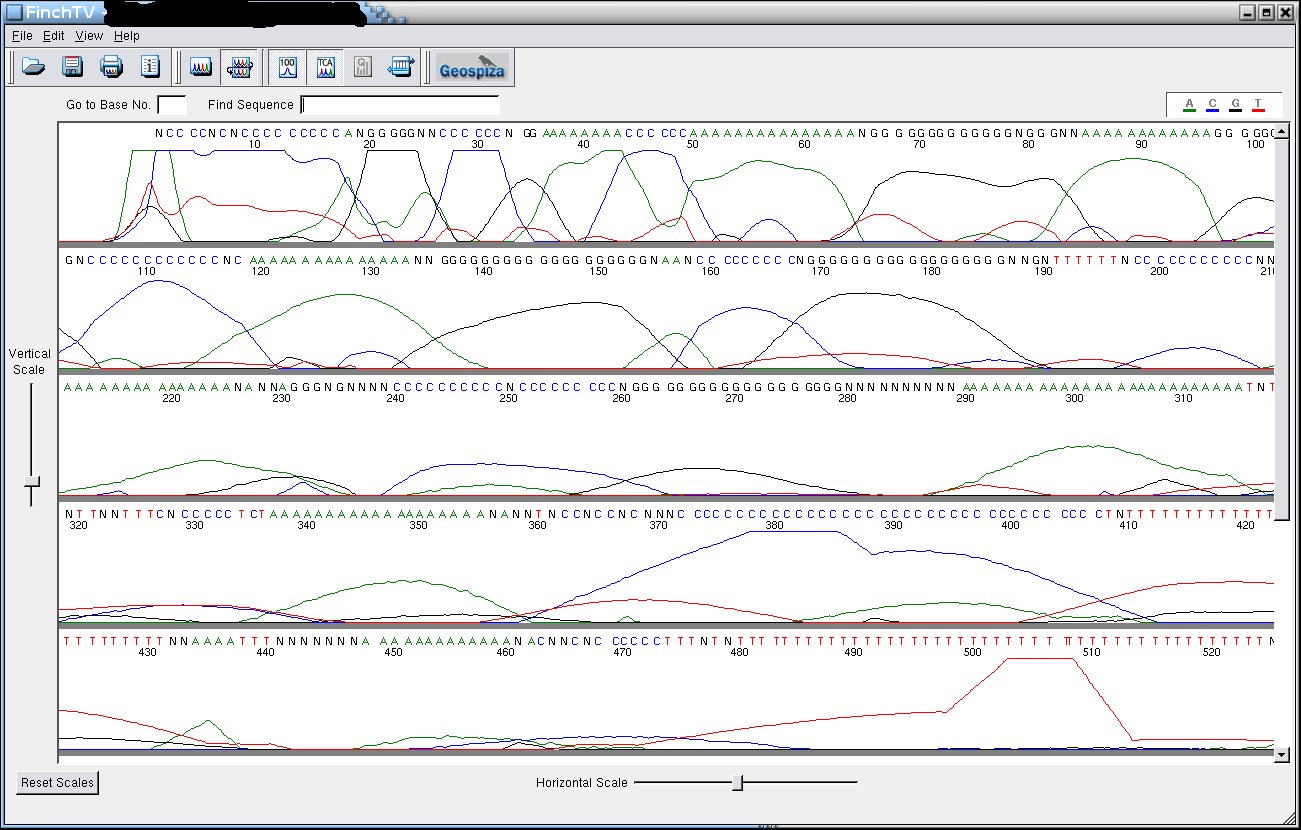
Autrement
D'autres
problèmes sont connus, mais je n'ai pas d'images pour l'instant.
Des séquences dont la qualité se dégrade
rapidement sont souvent dues à une mauvaise qualité de
matrice (contamination par des sels ou autre chose). Les suites d'un
même nucléotide peuvent poser un problème pour la
polymérase, qui patine dessus et qui en sort totalement
décalée. Ceci se traduit souvent par des vagues de
couleur sans séquence interprétable. Le cas type est le
poly-A des ADNc (vu comme poly-T dans les séquences). D'autres
séquences peuvent poser problème, comme des
séquences riches en GC, particulièrement quand celles-ci
peuvent former des structures secondaires. Le résultat est une
séquence parfaitement normale mais qui s'arrête
brusquement ou qui continue avec des signaux nettement plus faibles.
Finalement, le séquençage est une
suite très longue de manipulations. Il peut y avoir un
problème lors de la préparation de la réaction, de
la récupération, de la précipitation, de
remplissage de capillaires, de migration dans le capillaire. Une seule
solution si vous êtes sûr
que votre prep est bonne: recommencer le séquençage.
Et si, avec tout ça, ça ne marche toujours pas, il ne
reste plus qu'à partir très loin, en Chine ou à
Arcachon.
Acknowledgements
Je tiens à remercier les personnes (y compris moi-même)
qui, sans le savoir (sauf moi-même), ont contribué
à illustrer cette page et sans qui on rigolerait moins en
séquençage, ce qui serait bien dommage, sans blague,
m'enfin ...








